Développement d’un réseau de neurone entrainé à partir de tables de k-mer pantranscriptomique pour applications en RNA-Seq
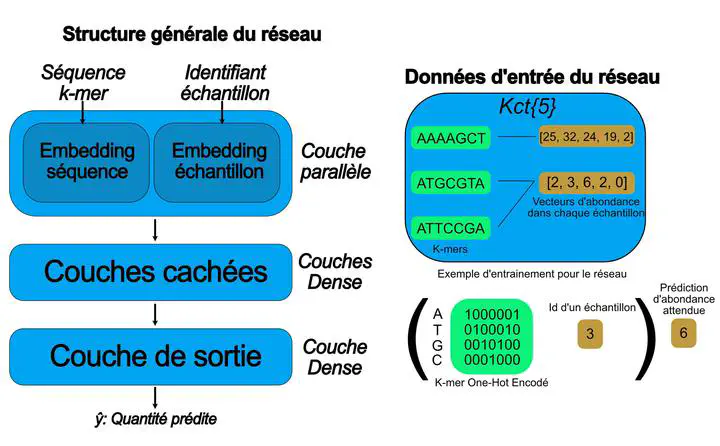
Bien que les réseaux de neurones soient capables de produire des embeddings de données d’expression génétiques, ce type de données s’appuie sur un alignement des séquences avec une référence, impliquant une perte d’information avec les séquences (bien qu’exprimées) n’alignant pas sur la référence. Entrainer à partir des lectures du RNA-Seq, divisées en k-mers, implique cependant un défi lié à la dimensionalité des données. Bien que les k-mers devraient être plus riches en information, elle est difficile à extraire de par le bruit et la dimensionalité de ce type de données. Générer un embedding représentatif de profils transcriptomiques à partir de ces données pourrait non seulement avoir une capacité prédictive au moins équivalente à profil classique, voir supérieur de par la richesse de ces données, mais aussi montrerait que des très courtes séquences contiennent suffisamment d’information pour l’assemblage d’une tel profil et permettrait potentiellement de faire du séquençage avec de très courtes séquences.