Utilisation de modèles de diffusion d’apprentissage profond pour débruiter les données de séquençage d’ARN à faible couverture
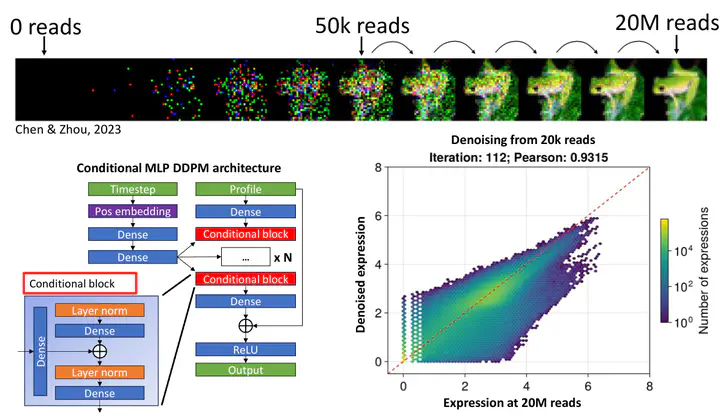
Bien que le séquençage d’ARN (RNA-seq) nous permette d’avoir une connaissance plus profonde de la biologie humaine, la quantité d’échantillons qui peuvent être séquencés est toujours limitée par les coûts de séquençage. Le coût par échantillon peut être réduit en diminuant la profondeur de séquençage, mais cela mène à des données de moindre qualité. De plus, les techniques existantes pour artificiellement augmenter la qualité des données, telles que les méthodes d’imputation dans le RNA-seq de single-cell, ne sont pas aptes pour débruiter données de RNA-seq standard (bulk RNA-seq).
Un modèle qui a le potentiel de résoudre ce problème est le modèle de diffusion. Nous voulons modifier ce modèle, créé à l’origine pour générer itérativement des images synthétiques, en liant les itérations du modèle à la profondeur de séquençage. Ceci permettrait de débruiter des données de RNA-seq de manière à complètement récupérer toute l’information biologique présente à pleine couverture, tel que le type d’échantillon ou les gènes différentiellement exprimés.
Ce modèle a le potentiel de créer de nouveaux standards de séquençage pour le RNA-seq dans de divers domaines expérimentaux ou hospitaliers, permettant à la fois de réduire les coûts et de procurer des grandes quantités de données.